
在实际的聚类应用中,通常使用k-均值和k-中心化算法来进行聚类分析,这两种算法都需要输入簇数,为了保证聚类的质量,应该首先确定最佳的簇数,并使用轮廓系数来评估聚类的结果。
通常情况下,使用肘方法(elbow)以确定聚类的最佳的簇数,肘方法之所以是有效的,是基于以下观察:增加簇数有助于降低每个簇的簇内方差之和,给定k>0,计算簇内方差和var(k),绘制var关于k的曲线,曲线的第一个(或最显著的)拐点暗示正确的簇数。
1,使用sjc.elbow()函数计算肘值
sjPlot包中sjc.elbow()函数实现了肘方法,用于计算k-均值聚类分析的肘值,以确定最佳的簇数:
library(sjPlot) sjc.elbow(data, steps = 15, show.diff = FALSE)
参数注释:
sjc.elbow()函数用于绘制k-均值聚类分析的肘值,该函数在指定的数据框计算k-均值聚类分析,产生两个图形:一个图形具有不同的肘值,另一个图形是连接y轴上的每个“步”,即在相邻的肘值之间绘制连线,第二个图中曲线的拐点可能暗示“正确的”簇数。
绘制k均值聚类分析的肘部值。 该函数计算所提供的数据帧上的k均值聚类分析,并产生两个图:一个具有不同的肘值,另一个图绘制在y轴上的每个“步”(即在肘值之间)之间的差异。 第二个图的增加可能表明肘部标准。
library(effects) library(sjPlot) library(ggplot2) sjc.elbow(data,show.diff = FALSE)
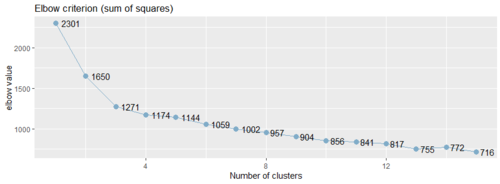
从下面的肘值图中,可以看出曲线的拐点大致在5附近:
2,使用NbClust()函数来验证肘值
从上面肘值图中,可以看到曲线的拐点是3,还可以使用NbClust包种的NbClust()函数,默认情况下,该函数提供了26个不同的指标来帮助确定簇的最终数目。
NbClust(data = NULL, diss = NULL, distance = "euclidean", min.nc = 2, max.nc = 15, method = NULL, index = "all", alphaBeale = 0.1)
参数注释:
利用NbClust()函数来确定k-均值聚类的最佳簇数:
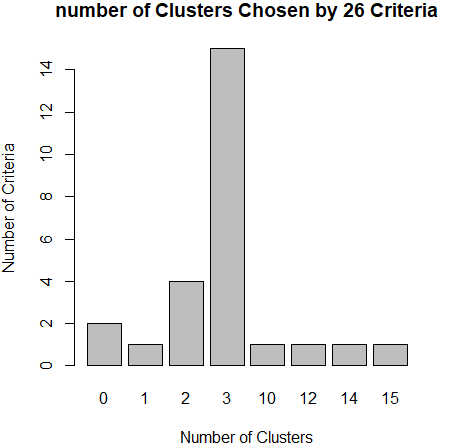
library(NbClust) nc <- NbClust(data,min.nc = 2,max.nc = 15,method = "kmeans") barplot(table(nc$Best.nc[1,]),xlab="Number of Clusters",ylab="Number of Criteria",main="number of Clusters Chosen by 26 Criteria")
从条形图种,可以看到支持簇数为3的指标(Criteria)的数量是最多的,因此,基本上可以确定,k-均值聚类的簇数目是3。
k-中心化聚类有两种实现方法,PAM和CLARA,PAM适合在小型数据集上运行,CLARA算法基于抽样,不考虑整个数据集,而是使用数据集的一个随机样本,然后使用PAM方法计算样本的最佳中心点。
通过fpc包中的pamk()函数得到最佳簇数:
pamk(data,krange=2:10,criterion="asw", usepam=TRUE, scaling=FALSE, alpha=0.001, diss=inherits(data, "dist"), critout=FALSE, ns=10, seed=NULL, ...)
参数注释:
使用pamk()函数获得PAM或CLARA聚类的最佳簇数:
library(fpc) pamk.best <- pamk(dataset) pamk.best$nc
通过cluster包中的clusplot()函数来查看聚类的结果:
library(cluster) clusplot(pam(dataset, pamk.best$nc))
三,评估聚类的质量(轮廓系数)
使用数据集中对象之间的相似性度量来评估聚类的质量,轮廓系数(silhouette coefficient)就是这种相似性度量,是簇的密集与分散程度的评价指标。轮廓系数的值在-1和1之间,该值越接近于1,簇越紧凑,聚类越好。当轮廓系数接近1时,簇内紧凑,并远离其他簇。
如果轮廓系数sil 接近1,则说明样本聚类合理;如果轮廓系数sil 接近-1,则说明样本i更应该分类到另外的簇;如果轮廓系数sil 近似为0,则说明样本i在两个簇的边界上。所有样本的轮廓系数 sil的均值称为聚类结果的轮廓系数,是该聚类是否合理、有效的度量。
1,fpc包
包fpc中实现了计算聚类后的一些评价指标,其中就包括了轮廓系数:avg.silwidth(平均的轮廓宽度)
library(fpc) result <- kmeans(data,k) stats <- cluster.stats(dist(data)^2, result$cluster) sli <- stats$avg.silwidth
2,silhouette()函数
包cluster中计算轮廓系数的函数silhouette(),返回聚类的平均轮廓宽度:
silhouette(x, dist, dmatrix, ...)
参数注释:
使用silhouette()计算轮廓系数:

library (cluster) library (vegan) #pam dis <- vegdist(data) res <- pam(dis,3) sil <- silhouette (res$clustering,dis) #kmeans dis <- dist(data)^2res <- kmeans(data,3) sil <- silhouette (res$cluster, dis)

聚类的结果,可以试用ggplot2来可视化,还可以使用的一些聚类包中特有的函数来实现:factoextra包,sjPlot包和cluster包
1,cluster包
clusplot()函数
2,sjPlot包
sjc.qclus()函数
该包中的两个函数十分有用,一个用于确定最佳的簇数,一个用于可视化聚类的结果。
(1),确定最佳的簇数fviz_nbclust()
函数fviz_nbclust(),用于划分聚类分析中,使用轮廓系数,WSS(簇内平方误差和)确定和可视化最佳的簇数
fviz_nbclust(x, FUNcluster = NULL, method = c("silhouette", "wss",), diss = NULL, k.max = 10, ...)参数注释:
例如,使用kmenas进行聚类分析,使用平均轮廓宽度来评估聚类的簇数:
library(factoextra) fviz_nbclust(dataset, kmeans, method = "silhouette")
(2),可视化聚类的结果
fviz_cluster()函数用于可是化聚类的结果:

fviz_cluster(object, data = NULL, choose.vars = NULL, stand = TRUE,
axes = c(1, 2), geom = c("point", "text"), repel = FALSE,
show.clust.cent = TRUE, ellipse = TRUE, ellipse.type = "convex",
ellipse.level = 0.95, ellipse.alpha = 0.2, shape = NULL,
pointsize = 1.5, labelsize = 12, main = "Cluster plot", xlab = NULL,
ylab = NULL, outlier.color = "black", outlier.shape = 19,
ggtheme = theme_grey(), ...)
参数注释:
使用fviz_cluster()把聚类的结果显示出来:
km.res <- kmeans(dataset,3) fviz_cluster(km.res, data = dataset)
参考文档:
确定最佳聚类数目的10种方法
作者:悦光阴
出处:http://www.cnblogs.com/ljhdo/
本文版权归作者和博客园所有,欢迎转载,但未经作者同意,必须保留此段声明,且在文章页面醒目位置显示原文连接,否则保留追究法律责任的权利。